O desenvolvimento da tecnologia do DNA recombinante inaugura a era da Biotecnologia Moderna. Em 1973, Herbert Boyer, da Universidade da Califórnia em São Francisco, e Stanley Cohen, da Universidade de Stanford, relataram a construção de organismos funcionais que combinavam e replicavam informações genéticas de diferentes espécies. Seus experimentos demonstraram o potencial de uma nova tecnologia recombinante de DNA (primórdios da engenharia genética), esta ferramenta biotecnológica podia chegar nos mais variados campos da medicina, farmacologia, indústria e agricultura. Os diversos estudos focados nas natureza química do DNA criaram um ambiente favorável ao desenvolvimento de novas tecnologias, permitindo a criação de métodos diagnósticos, preventivos e terapêuticos[1].
dna-fingerprinting
No início da revolução instaurada pela tecnologia do DNA recombinante, um britânico, Alec Jeffreys, quase que de forma acidental identificou uma particularidade em sequências de nucleotídeos (DNA). Jeffreys em seus estudos interessava-se pelas diferenças genéticas entre as espécies, suas pesquisas na Universidade de Leicester concentravam-se no gene da mioglobina, ele constatou que um pequeno trecho de DNA se repetia continuamente. Bem como um fenômeno similar havia sido observado, em 1980, por Ray Whinte e Arlene Wyman, que, ao examinar “outro gene”, constataram que o número dessas repetições variam de indivíduo para indivíduo.
Jeffreys decidiu usar essa sequência como uma sonda marcada radioativamente, deste jeito ele poderia “caçar” a sequência por todo o genoma. A partir deste estudo, foi observado que o curto trecho de DNA repetitivo aparecia não somente no gene da mioglobina, mas espalhado por todo genoma. Embora a técnica tivesse detectado muitas sequências similares em inúmeras amostras de DNA, ainda restava tanta variação entre uma amostra e as demais que, mesmo naquelas extraídas de membros da mesma família, era possível distinguir um indivíduo do outro.

Alec Jeffreys, English molecular biologist and discoverer of DNA fingerprinting.
Nas palavras do próprio Jeffreys em artigo publicado na revista Nature em 1985,
“O perfil nos fornece uma fingerprinting (impressão digital) específica de cada indivíduo”. Mais tarde essa tecnologia ficaria conhecida como “Polimorfismo no Comprimento do Fragmento de Restrição” (restriction Fragment length polymorphisms) ou pela sigla em inglês (RFLP). Com o tempo, percebeu-se que essa tecnologia também poderia distinguir outros animais e vegetais, de forma a descobrir, até mesmo, como cada criatura está relacionada com as demais e outra listas de aplicações infindáveis.
Durante muito tempo, os RFLP foram usados como prova chave em casos criminais famosos, no entanto, os geneticistas eram constantemente contestados por advogados em relação a eficiência da técnica. Inevitavelmente alguns resultados poderiam ser de difícil interpretação e, com isso, a identificação genômica passa a ser contestada por motivos técnicos e legais. No início, quando a identificação genômica era realizada em laboratórios forenses não especializados, por profissionais com pouco entendimento sobre a natureza química do DNA, erros não eram incomuns (como contaminação de amostras). Os órgãos de segurança pública e entidades internacionais logo perceberam que, se quisessem que essa nova e poderosa tecnologia continuasse sendo usada, tais problemáticas teriam que ser resolvidas[2].
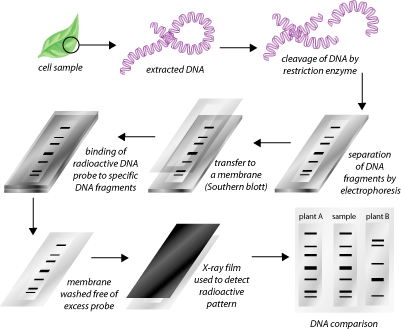
Desenho esquemático de como é obtido o DNA fingerprint. Inicialmente é feita a coleta da amostra, seguida de extração e clivagem do DNA. Depois da clivagem o material é separado por eletroforese seguida de marcação radioativa, finalizando com a comparação de fragmentos e obtenção da “impressão digital”.
Foi quando um novo marcador genético, as chamadas “repetições curtas enfileiradas” (short tandem repeat) conhecidas pela sigla em inglês STRs, substituíram o método usando os RFLPs. Os tamanhos do STRs podiam ser medido com bastante precisão, eliminando-se a avaliação subjetiva dos RFLPs. No entanto podemos encontrar em uma população alelos iguais de uma mesma sequência entre indivíduos, onde a diferença entre eles está apenas no número de vezes que a unidade se repete. Assim, estas sequências são altamente polimórficas (variam de indivíduo para indivíduo).
Um exemplo: a sequência ATCG repete-se de modo consecutivo 3 vezes, obtendo portanto, no alelo onde ela se localiza, a seguinte sequência ATCGATCGATCG, originando um fragmento de 12 pares de bases. Agora, se pensarmos que essa sequência se repetia 4 vezes no alelo, obteríamos um fragmento como este ATCGATCGATCGATC, com 15 pares de bases. Estes dois alelos podem ser diferenciados de acordo com seus respectivos tamanhos. São justamente estas diferenças no número de repetições das sequências que resultam na detecção de diferentes alelos, ou seja, fragmentos de tamanhos diferentes no genoma de diferentes indivíduos.
Com o passar dos anos e as novas tecnologias de sequenciamento, foi possível melhorar os testes de identificação genéticos. Ao mesmo tempo, novas estratégias foram desenvolvidas como o polimorfismo de um único nucleotídeo, do inglês Single-Nucleotide Polymorphism (SNP). Como o próprio nome indica, é detectada a repetição de um único par de nucleotídeos. Conforme o entendimento da comunidade científica for aumentando em relação a natureza química e funcional da molécula de DNA, ela passará a ter mais notoriedade em outras áreas do conhecimento. No sistema do Departamento Federal de Investigação Estadunidense (FBI), por exemplo, a identificação genômica pode fazer parte de um processo de investigação criminal ou mesmo de paternidade. Todavia qualquer teste diagnóstico de cunho científico está em constante evolução.
Quando imaginamos uma população de milhões de pessoas em um mundo globalizado, a possibilidade de existir dois indivíduos diferentes em uma mesma população, com a mesma sequência, mesmo número de repetições na mesma região do genoma não é tão utópica. Portanto, é necessário examinar diversas regiões, de forma que, quanto maior o número de locais analisado no genoma, menor seja a probabilidade de uma mesma amostra corresponder a duas pessoas diferentes. Até o ano de 2016, o FBI realizava análise de treze regiões e de um marcador que determina o sexo do indivíduo de quem a amostra de DNA fora obtida [3].
No Brasil, o Ministério do Trabalho descreve os profissionais em biotecnologia como profissionais de nível superior aptos na manipulação de material genético, na síntese de sequências de DNA, na análise de genes e genomas, na identificação de marcadores genéticos, dentre outros. De acordo com essas atribuições, o conhecimento deste profissional tem muito a contribuir em instituições e órgão de pesquisa, ou investigação, de características genéticas, atuando como profissional da polícia científica e/ou perícia criminal[4].
Gostaria de saber mais sobre o DNA fingerprinting ? Comente abaixo, vamos construir conhecimento juntos!


Bibliografia
[1] http://www.genomenewsnetwork.org/resources/timeline/1973_Boyer.php
[2] Livro: DNA o segredo da vida, editora; companhia das letras, autor; James D. Watson.
[3] http://genetica-humana.blogspot.com.br/p/genetica-forense.html
[4] http://www.mtecbo.gov.br/cbosite/pages/pesquisas/BuscaPorTituloResultado.jsf







Excelente conteúdo!
Estou estudando para o concurso do IGP/RS, cargo de perito criminal, e este artigo veio bem a calhar.
Continue postando conteúdo de biotec forense, vai me ajudar nas revisões de conteúdo!
Se tiveres algum material específico me manda por e-mail.
Abraço
Olá Salus, ficamos felizes que tenha gostado de nosso conteúdo, na mesma linha temos o texto que fala sobre a biotecnologia ser utilizada para resolver crimes: (http://profissaobiotec.com.br/como-a-biotecnologia-pode-ajudar-a-resolver-crimes/).
Continue nos acompanhando e participando!
Boa sorte no concurso!